Posttranskripciona modifikacija
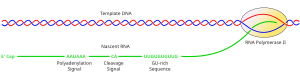
Posttranskripciona modifikacija ili kotranskripciona modifikacija je proces u eukariotskim ćelijama u kome se primarni transkript RNK konvertuje u zrelu RNK. Značajan primer je konverzija prekurzorske informacione RNK u krajnju informacionu RNK (iRNK) koja se formira pre translacije proteina. Proces obuhvata tri glavna koraka: adicija 5' kape, adicija 3' poli-adenilacionog repa, i splajsovanje. Ovaj proces je vitalan za korektnu translaciju genoma eukariota, zato što inicijalna prekurzorska iRNK koja se formira tokom transkripcije sadrži eksone (kodirajućie ili važne sekvence koje učestvuju u translaciji), i introne (nekodirajuće sekvence).[1]
Baze podataka
уреди| Ime | Opis | Tip | Link | Reference |
|---|---|---|---|---|
| RMBase | RMBase je dizajnirana za dekodiranje prostora RNK modifikacija identifikovanog na bazi sekvenciranja visokog kapaciteta (Pseudo-seq, Ψ-seq, CeU-seq, Aza-IP, MeRIP-seq, m6A-seq, miCLIP, m6A-CLIP, RiboMeth-seq). U ovom izvoru su demonstrirane hiljade RNK modifikacija lociranih na iRNK, regulatornim ncRNK (e.g. lncRNA, miRNA), mesta vezivanja miRNA i SNP mutacije vazane za bolesti. | baza podataka | vebsajt[мртва веза] | [2] |
| MODOMICS | MODOMICS je baza podataka RNK modifikacija koja pruža sveobuhvatne informacije o hemijskim strukturama modifikovanih ribonukleozida, nihovim biosintetičkim putevima, enzimima koji modifikuju RNK i lokacijama modifikovanih ostataka u RNK sekvencama. | baza podataka | vebsajt | [3] |
| RNAMDB | RNAMDB služi kao fokalna tačka za informacije vezane za prirodne RNK modifikacije | baza podataka | vebsajt | [4] |
| . | ||||
Vidi još
уредиReference
уреди- ^ Berg, Tymoczko & Stryer 2007, стр. 836
- ^ Sun, WJ; Li, JH; Liu, S; Wu, J; Zhou, H; Qu, LH; Yang, JH (11. 10. 2015). „RMBase: a resource for decoding the landscape of RNA modifications from high-throughput sequencing data.”. Nucleic Acids Research. 44: gkv1036. PMC 4702777 . PMID 26464443. doi:10.1093/nar/gkv1036.
- ^ Machnicka, MA; Milanowska, K; Osman Oglou, O; Purta, E; Kurkowska, M; Olchowik, A; Januszewski, W; Kalinowski, S; Dunin-Horkawicz, S; Rother, KM; Helm, M; Bujnicki, JM; Grosjean, H (decembar 2012). „MODOMICS: a database of RNA modification pathways--2013 update.”. Nucleic Acids Research. 41 (Database issue): D262—7. PMC 3531130 . PMID 23118484. doi:10.1093/nar/gks1007.
- ^ Cantara, WA; Crain, PF; Rozenski, J; McCloskey, JA; Harris, KA; Zhang, X; Vendeix, FA; Fabris, D; Agris, PF (decembar 2010). „The RNA Modification Database, RNAMDB: 2011 update.”. Nucleic Acids Research. 39 (Database issue): D195—201. PMC 3013656 . PMID 21071406. doi:10.1093/nar/gkq1028.
Literatura
уреди- Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2007), Biochemistry (6 изд.), New York: WH Freeman & Co., ISBN 978-0-7167-6766-4
- Hames, David; Hooper, Nigel (2006), Instant Notes Biochemistry (3 изд.), Leeds: Taylor and Francis, ISBN 978-0-415-36778-3
Spoljašnje veze
уредиRMBase RNA Modification Base is designed for decoding the landscape of RNA modifications identified from high-throughput sequencing data (Pseudo-seq, Ψ-seq, CeU-seq, Aza-IP, MeRIP-seq, m6A-seq, miCLIP, m6A-CLIP, RiboMeth-seq).
- Post-Transcriptional+RNA+Modification на US National Library of Medicine Medical Subject Headings (MeSH)